Distanza genetica tra le popolazioni
Nell'articolo del 1967 di Luigi Luca Cavalli-Sforza and Anthony W. F. Edwards "Phylogenetic analysis: models and estimation procedures" apparso sulla rivista American Journal of Human Genetics, si ricorre al prodotto scalare di vettori per misurare la "distanza genetica" tra le popolazioni.
Dell'interessante articolo riportiamo solo la parte relativa all'uso del prodotto scalare di vettori, rimandando i lettori interessati alla lettura integrale dello stesso per ulteriori approfondimenti.
Per il loro esperimento gli autori hanno scelto elementi delle quattro popolazioni (indicate nella tabella sottostante) e di ogni gruppo hanno determinato le frequenze fin dei geni nei gruppi di sangue A B 0.
I dati in tabella riportano la distinzione tra quattro alleli A1, A2, B, O.
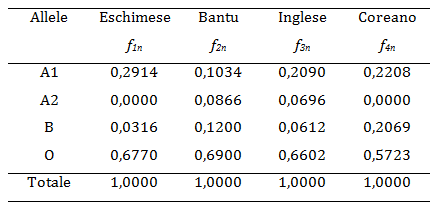
Per calcolare la "distanza" hanno fatto ricorso al prodotto di vettori secondo lo schema seguente.
Si costruiscono i vettori normalizzati e, siccome stiamo lavorando con le frequenze, è sufficiente eseguire le radici quadrate dei valori presenti nella tabella.
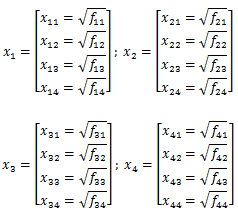
Per poterli confrontare fra loro li disponiamo su una sfera (in uno spazio quadrimensionale) di raggio unitario (l'immagine successiva simula il procedimento da seguire nel caso gli alleli fossero tre).
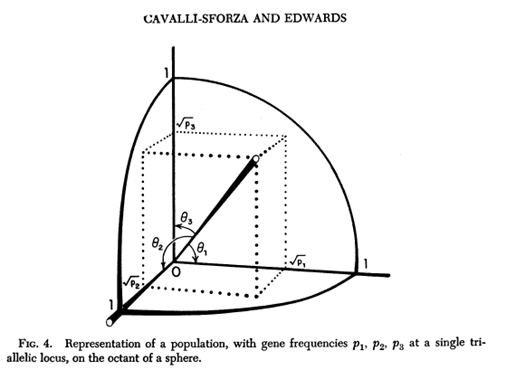
Figura tratta dall'articolo citato, visualizza una situazione in uno spazio a tre dimensioni
Se ora moltiplichiamo i vettori
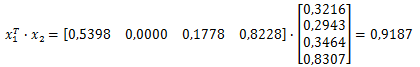
otteniamo il valore coseno dell’angolo che si forma tra i due vettori considerati:
cos(α12 )=0,9187 da cui α12=α21=arcos(0,9187)=0,4062 rad
Seguendo l'immagine possiamo prendere questo valore come indicatore della "distanza genetica" tra la popolazione Eschimese e quella Bantu.
Ora ripetiamo i calcoli per determinare gli altri quattro valori.
Possiamo scrivere la seguente matrice simmetrica:
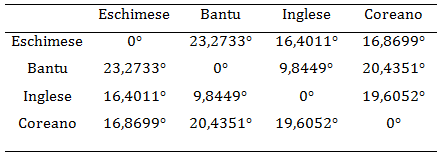
Leggendo i valori possiamo osservare che la più grande "distanza genetica" si ha tra la popolazione Eschimese e quella Bantu; mentre quella più breve è quella tra le popolazioni Bantu e Inglesi.
